
实验三十一 微生物基因组测序与序列组装
一、实验目的
(1)了解下一代测序技术的原理及其在微生物学研究中的应用。
(2)掌握基因组装软件Velvet的使用方法。
二、实验原理
第一代测序技术始于1975年Sanger的双脱氧链终止法,发展到现在,Sanger测序用四种不同的荧光染料分别标记片段末端不同的碱基,通过电泳将不同长度的片段分开,根据末端碱基得到原始序列信息。目前,Sanger测序可以测到800~1 000个碱基,但是测序通量很小,而且价格昂贵。2004—2005年间开始商业化使用的第二代测序技术(Next-Generation Sequencing)克服了以上两个缺点,它可以同时对多个DNA片段进行平行测序:将打碎后建库的DNA片段锚定在固体介质表面,比如通过连接接头的方法将DNA片段锚定在多个磁珠上进行PCR反应(Roche/454平台),或者锚定在测序通道内表面进行桥式PCR(Illumina平台)。通过对每个锚定DNA每加一个碱基进行一次“加上荧光染料—洗脱多余染料—荧光成像扫描”的循环过程,实现平行高通量的深度测序(图30-1)。目前常用的平台是Roche/454公司的FLX测序仪Illumina的HiSeq 2000测序仪和ABI的SOLiD测序平台。根据提供的DNA来源和前期处理的不同,二代测序技术可以用以解答不同研究目的的生物学问题,如可以用于微生物研究中的比较基因组学、转录组学、宏基因组学等。
本实验将学习使用Velvet软件组装Illumina/Solexa平台基因组测序结果。
Velvet软件主要有两个程序组成:velveth和velvetg。
(1)velveth的输入默认是fasta格式的序列文件,也能识别fastq、fasta.gz、fastq.gz、sam、bam、eland和gerald文件。序列类型默认是short,也可以是shortPaired、short2、shortPaired2、long或longPaired。
命令格式为:
$./velveth output_directory hash_length[[-file_format][-read_type]filename]
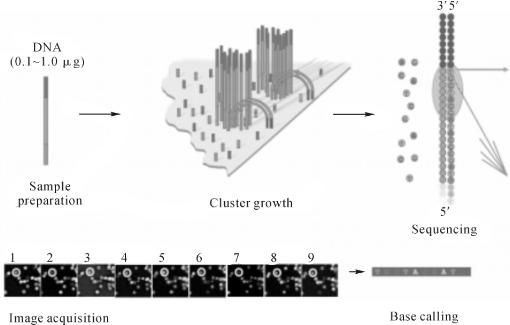
图31-1 Illumina测序原理
velveth运行的结果生成一个hash表,并输出3个文件,其中Roadmaps和Sequences文件是下一步velvetg程序运行必需的。
Log:日志文件
Roadmaps:路线图文件
Sequences:序列文件,包含所有输入的序列
(2)velvetg是velvet的核心程序,其命令格式为:
$./velvetg output_directory/[-cov_cutoff][-max_coverage]…
运行的结果输出以下文件:
contigs.fa:fasta格式的组装好的片段,长度大于2k(k为velveth运行时用的字长)
PreGraph:中间组装图
LastGraph:最后组装图
Graph:最后组装图
stats.txt:统计信息
三、实验材料与仪器
(1)计算机(安装有Ubuntu Linux系统)。
(2)E.coli基因组测序原始序列文件,E.coli K12的基因组测序数据可以从下面网址下载:http://download.clcbio.com/testdata/raw_data/solexa.zip。
四、实验步骤
(1)分离细菌E.coli单克隆,菌株在25mL LB中培养过夜,用于基因组DNA提取。
(2)基因组提取可以用细菌基因组提取试剂盒,如QIAGEN DNeasy Blood &Tissue Kit,DNA提取步骤参考试剂盒说明手册。
(3)紫外光谱检测提取的基因组DNA质量。一般基因组DNA样品(~20μg)在230nm与260nm有吸收峰,要求比值280/260>1.8;并且比值260/230 >2。
(4)每菌株样品提交至少2μg基因组DNA用于高通量测序。目前测序公司常见用Illumina公司的HiSeq2000测序仪,可测两末端各100bp的数据。测序文库的构建流程及其他Illumina平台测序技术可以参考Illumina公司网站的说明:http://www.illumina.com/technology/sequencing_technology.ilmn。
(5)测序数据的预处理。高通量测序的序列数据一般存储在FASTQ格式文件,文件后缀一般为.fastq,.fq等。FASTQ格式以每个测序读长(read)为4行,分别为头、序列、序列ID(可选)和质量分数(ASCII编码表示)。
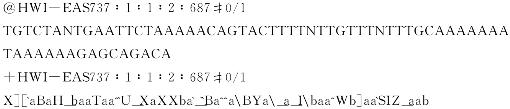
(6)测序原始数据可以用FastQC评价质量好坏。FastQC可以从网址(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)下载。运行FastQC图形界面,打开FASTQ数据文件,就会以显示质量报告。检查per-base quality,persequence quality及per-base content等。其中overrepresentation of sequences可能是测序PCR假阳性(artifacts)。通过参考FastQC图标的颜色判断质量好坏(绿色代表正常;橙色代表可能有些问题;红色代表非常可能有问题)。但是也要注意测序中有些小的异常是可以接受的,并不会对后续数据分析造成影响。
(7)如果数据质量有问题,可以通过FASTX-Toolkit软件对数据文件进行处理,如一般要求测序数据的reads质量(-q)都为20以上:
$fastq_quality_filter-Q33-q 20-p 80-i infile.fq-o outfile.fq
[-q N]代表只保留最小质量分数N以上。
[-p N]代表具有以上-q质量的碱基占的最小百分率。
其他命令可以参考网站说明http://hannonlab.cshl.edu/fastx_toolkit。
(8)Velvet组装基因组。
①下载Velvet:http://www.ebi.ac.uk/~zerbino/velvet/。
②Velvet软件编译:
$cd
$mkdir assemble_velvet
$cp/home/bioinfo/Downloads/velvet_1.2.10.tgz assemble_velvet/
$cd assemble_velvet
$tar zxvf velvet_1.2.10.tgz#解压
$cd velvet_1.2.10
$make #编译
$sudo apt-get install velvet #如果上面编译不成功可以用这条命令安装
③首先利用velvet自带的脚本程序对每一个pair-end数据进行合并:
$shuffleSequences_fastq.pl s1_1.fq s1_2.fq s1.fq
④运行velveth格式化reads:
$velveth assembly_all 23-fastq-shortPaired s1_paired.fq-short s1_1.fq-unique.outshort s1_2.fq-unique.out
这里哈希长度(K-mer)为23,输入文件格式为fastq(-fastq),测序reads类型分别为配对的双末端序列(-shortPaired)与末配对的单端序列(-short)。
*K-mer值必须为奇数,且小于MAXKMERLENGTH,这个值默认为31,test multiple K-mer values,and calculate the total number of contigs,N50,and N90for each assembly.
⑤运行velvetg组装序列:
$velvetg assembly_all-cov_cutoff auto-exp_cov auto-ins_length500-ins_length_sd50
这里assembly_all是工作目录
-ins_length:双端测序reads中间插入片段的长度
-cov_cutoff:过滤覆盖度域值,默认不移除
-exp_cov:测序区域的期望覆盖率,auto为程序自动
-ins_length_sd:数据集的标准差,默认corresponding length的10%
上述组装命令运行后,会产生拼装得到的序列,为组装重叠群contigs,存储在工作目录下contigs.fa文件中。
⑥检查序列组装(assembly)结果:
$count_fasta.pl assembly_all/contigs.fa
根据出来的N50和max contig长度来判断拼接的效果,contig数尽量小。可以改变选项和参数,得到最优结果。最后组装得到的Congtigs序列可以用于后续基因组分析与实验验证,如基因预测、比对基因组等。
五、实验报告
(1)运行环境(包括操作系统和软件),实验步骤,结果文件记录。
(2)上机实验中遇到的问题及其解决方法。
六、思考题
(1)下一代测序技术有哪些?其中Illumina平台的测序原理是什么?
(2)基因组组装的参数N50代表什么?
参考文献
[1]Metzker M L.Sequencing technologies—the next generation[J].Nature Review of Genetics.2010,11(1):31—46.
[2]Royce L,Boggess E,Jin T,et al.Identification of Mutations in Evolved Bacterial Genomes.In:Alper HS,editor.Systems Metabolic Engineering[M].Humana Press,2013:249—267.
[3]秦楠,栗东芳,杨瑞馥.高通量测序技术及其在微生物学研究中的应用[J].微生物学报,2011,51(4):445—457.
上一篇:褶皱背斜和向斜示意图
下一篇:施工方案和施工组织设计审批程序







